번역 (생물학)
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
번역(생물학)은 리보솜에 의해 mRNA의 유전 정보를 바탕으로 단백질을 합성하는 과정을 의미한다. 리보솜은 mRNA의 코돈에 상보적인 tRNA를 통해 아미노산을 연결하여 폴리펩타이드 사슬을 형성하며, 이러한 과정은 개시, 신장, 종결의 세 단계로 이루어진다. 번역은 세포 내에서 일어나는 복잡한 과정으로, 오류가 발생하거나 조절될 수 있으며, 암과 같은 질병의 발생과도 관련이 있다. 번역 과정은 수학적 모델로 연구되며, 유전 부호는 특정 코돈이 아미노산을 지정하는 규칙을 제공한다.
더 읽어볼만한 페이지
- 단백질 생합성 - 리보솜
리보솜은 rRNA와 단백질로 구성된 두 개의 소단위체로 이루어진 세포 내 단백질 합성 복합체로서, 세포질 내에서 mRNA의 코돈을 해독하여 폴리펩타이드 사슬을 합성하는 번역 과정을 수행하며, 원핵세포와 진핵세포의 구조적 차이로 인해 항생제 개발의 표적이 되고, RNA 세계에서 기원한 자기 복제 복합체로 추정된다. - 단백질 생합성 - 종결 코돈
종결 코돈은 표준 유전 암호에서 단백질 합성을 종료시키는 역할을 하는 UAG(앰버), UAA(오커), UGA(오팔 또는 엄버) 세 가지 코돈이며, 넌센스 돌연변이, 논스톱 돌연변이 등 다양한 유전 질환과 관련이 있을 수 있다. - 유전자 발현 - RNA 간섭
RNA 간섭은 이중가닥 RNA에 의해 유전자 발현이 억제되는 과정으로, 다이서 효소에 의해 생성된 작은 간섭 RNA가 RNA 유도 침묵 복합체와 결합하여 표적 mRNA를 분해하거나 번역을 억제하며, 바이러스 방어, 발생 조절, 유전체 안정성 유지 등 다양한 기능과 응용 분야를 가진다. - 유전자 발현 - 유전자 재조합
유전자 재조합은 유성 생식 생물의 염색체 유전자 교차를 통한 유전자 재배열 현상으로, 유전적 변이를 증가시키고 유전학적 지도 작성에 활용되며, 생명공학 등 다양한 분야에서 기술로 이용된다. - 분자생물학 - 단백질
단백질은 아미노산 중합체로 생체 구조 유지와 기능에 필수적이며, 아미노산 서열에 따라 고유한 3차원 구조를 형성하여 효소, 구조, 수송, 저장, 수축, 방어, 조절 단백질 등 다양한 기능을 수행하고, 인체 내에서 건강 유지와 질병 예방에 중요한 역할을 하는 필수 영양소이다. - 분자생물학 - 의학
의학은 질병의 진단, 예후, 치료, 예방을 연구하는 과학 및 실천 분야이며, 고대부터 발전하여 현대에는 다양한 전문 분야로 세분화되고 첨단 기술 발전에 따라 혁신적인 변화를 겪고 있다.
2. 번역의 기본 원리
번역은 전령 RNA(mRNA)의 유전 정보를 해독하여 아미노산을 순서대로 연결해 폴리펩타이드를 만드는 과정이다.
단백질 생성은 리보솜이 단백질의 C-말단에 아미노산을 하나씩 추가하면서 이루어진다.[1] 이때 추가되는 아미노산의 종류는 mRNA에 의해 결정된다. mRNA의 뉴클레오티드 삼중항(코돈)이 특정 아미노산을 지정하며, 이 코돈에 상보적인 안티코돈을 가진 전이 RNA(tRNA)가 해당 아미노산을 리보솜으로 운반한다.[3] mRNA의 코돈 서열에 따라 tRNA가 운반하는 아미노산이 순서대로 연결되면서 폴리펩타이드 사슬이 만들어진다.
mRNA는 염색체에서 리보솜으로 유전 정보를 전달하는 역할을 한다. 리보솜은 이 정보를 바탕으로 코돈을 "읽고", 각 코돈에 해당하는 아미노산을 가진 tRNA를 불러들인다. tRNA는 아미노산 부착 부위와 안티코돈 부위를 가지고 있으며, 아미노아실 tRNA 합성효소에 의해 특정 아미노산과 결합한다. 이렇게 아미노산과 결합한 tRNA는 "충전된" 상태가 되며, 박테리아에서는 EF-Tu에 의해 리보솜으로 운반된다.[6]
리보솜에는 tRNA가 결합하는 두 개의 부위 (A 부위와 P/E 부위)가 있다. A 부위는 mRNA의 코돈에 상보적인 안티코돈을 가진 tRNA가 결합하는 곳이고, P/E 부위는 이미 폴리펩타이드 사슬에 연결된 tRNA가 위치하는 곳이다. A 부위에 새로운 tRNA가 들어오면, P/E 부위에 있던 tRNA의 아미노산과 A 부위에 있는 tRNA의 아미노산 사이에 펩타이드 결합이 형성된다. 이후 폴리펩타이드 사슬은 A 부위의 tRNA로 이동하고, P/E 부위의 tRNA는 리보솜에서 떨어져 나간다. 이러한 과정이 반복되면서 폴리펩타이드 사슬이 길어진다.[7]
새로운 아미노산이 추가되고 tRNA가 리보솜에서 방출된 후에는, 전위 효소 (EF-G 또는 a/eEF-2)에 결합된 GTP의 가수분해 에너지를 이용하여 리보솜이 mRNA의 3' 말단 방향으로 한 코돈씩 이동한다.[8] 번역 속도는 원핵생물에서 진핵생물보다 빠르다.[9]
번역은 다음 3단계로 진행된다.
1. '''개시''': 표적 mRNA 주위에 리보솜이 모인다. 첫 번째 tRNA가 mRNA의 개시 코돈에 결합한다.
2. '''신장''': 리보솜 소단위체에 의해 확인된(''수용'') tRNA는 운반한 아미노산을 리보솜 대단위체로 옮기고, 그 전에 수용된 아미노산에 결합한다(''펩티드 전이''). 그 후, 리보솜은 다음 mRNA 코돈으로 이동하여 과정을 계속하고(''전좌''), 아미노산 사슬을 형성한다.
3. '''종결''': 종결 코돈에 도달하면 리보솜은 폴리펩타이드를 방출한다. 리보솜 복합체는 그대로 남아, 다음에 번역될 mRNA로 이동한다.
원핵생물(세균 및 고세균)은 세포질 기질에서, 진핵생물은 세포질 내 또는 소포체 막을 넘어 공동 번역 전위 과정을 통해 번역이 일어난다.
2. 1. 리보솜의 역할
리보솜은 단백질 합성이 일어나는 세포 내 작은 기관이다.[1] 리보솜은 작은 소단위체와 큰 소단위체, 두 개의 소단위체로 구성된다. 이 소단위체는 전령 RNA(mRNA)를 단백질로 번역하기 전에 함께 모여서 번역이 수행될 장소와 폴리펩타이드가 생성될 장소를 제공한다.[2] 추가할 아미노산 유형은 mRNA 분자에 의해 결정된다. 추가되는 각 아미노산은 mRNA의 3개의 뉴클레오티드 서브시퀀스(코돈)에 매칭된다. 사슬에 추가된 연속적인 아미노산은 mRNA의 연속적인 뉴클레오티드 삼중항에 매칭된다. 이러한 방식으로 템플릿 mRNA 사슬의 뉴클레오티드 서열은 생성된 아미노산 사슬의 아미노산 서열을 결정한다.[3] 아미노산의 추가는 펩타이드의 C-말단에서 발생하며, 따라서 번역은 아민에서 카르복실 방향으로 진행된다.[4]mRNA는 염색체에서 리보솜으로 리보뉴클레오티드 서열로 인코딩된 유전 정보를 전달한다. 리보뉴클레오티드는 코돈이라고 하는 일련의 뉴클레오티드 삼중항에서 번역 기계에 의해 "읽혀진다". 각 삼중항은 특정 아미노산을 암호화한다.
리보솜은 리보솜 RNA(rRNA)와 단백질을 포함하는 다중 소단위체 구조이며, 아미노산이 단백질로 조립되는 "공장"이다.
전령 RNA(tRNA)는 아미노산을 리보솜으로 수송하는 작은 비코딩 RNA 사슬(74–93 뉴클레오티드)이다. tRNA는 아미노산 부착 부위와 안티코돈을 갖는다. 안티코돈은 그들의 화물인 아미노산을 암호화하는 mRNA 삼중항에 상보적인 RNA 삼중항이다.
아미노아실 tRNA 합성효소는 특정 tRNA와 그 안티코돈 서열이 요구하는 아미노산 간의 결합을 촉매한다. 이 반응의 생성물은 아미노아실-tRNA이다. 아미노산은 카르복실기를 통해 3' OH의 tRNA에 에스터 결합으로 결합한다. tRNA에 아미노산이 연결되면 tRNA는 "충전"된 것으로 간주된다. 박테리아에서 이 아미노아실-tRNA는 EF-Tu에 의해 리보솜으로 운반되며, 여기서 mRNA 코돈은 특정 tRNA 안티코돈에 상보적인 염기쌍을 통해 일치한다. tRNA를 잘못된 아미노산과 잘못 짝지은 아미노아실-tRNA 합성효소는 잘못 충전된 아미노아실-tRNA를 생성하여 단백질의 해당 위치에 부적절한 아미노산을 생성할 수 있다.
리보솜에는 tRNA에 대한 두 개의 결합 부위가 있는데, 아미노아실 부위(A)와 펩티딜 부위/출구 부위(P/E)이다. mRNA와 관련하여 세 부위는 5'에서 3' E-P-A로 정렬되는데, 이는 리보솜이 mRNA의 3' 말단을 향해 이동하기 때문이다. A 부위는 mRNA의 상보적인 코돈이 있는 들어오는 tRNA에 결합한다. P/E 부위는 성장하는 폴리펩타이드 사슬이 있는 tRNA를 유지한다. 아미노아실-tRNA가 처음에 mRNA의 해당 코돈에 결합하면 A 부위에 있다. 그런 다음 A 부위의 tRNA 아미노산과 P/E 부위에 충전된 tRNA의 아미노산 사이에 펩티드 결합이 형성된다. 성장하는 폴리펩타이드 사슬은 A 부위의 tRNA로 전달된다. 전위가 발생하여 tRNA를 아미노산이 없는 P/E 부위로 이동시킨다. 폴리펩타이드 사슬로 이제 충전된 A 부위에 있던 tRNA는 P/E 부위로 이동하고 비충전 tRNA는 떠나고 다른 아미노아실-tRNA가 A 부위에 들어가서 과정을 반복한다.[7]
새로운 아미노산이 사슬에 추가된 후, tRNA가 리보솜 밖으로 방출되어 세포질로 들어간 후, 전위 효소 EF-G(세균) 및 a/eEF-2(진핵생물 및 고세균)에 결합된 GTP의 가수분해에 의해 제공되는 에너지는 리보솜을 3' 말단을 향해 한 코돈씩 이동시킨다. 단백질의 번역에 필요한 에너지는 상당하다. ''n''개의 아미노산을 포함하는 단백질의 경우, 번역에 필요한 고에너지 인산 결합의 수는 4''n''-1이다.[8] 번역 속도는 다양하며, 진핵 세포(초당 최대 6~9개의 아미노산 잔기)보다 원핵 세포에서 훨씬 높다(초당 최대 17~21개의 아미노산 잔기).[9]
2. 2. tRNA의 역할
전령 RNA(mRNA)는 아미노산을 리보솜으로 운반하는 작은 비암호화 RNA 사슬(74–93 뉴클레오타이드)이다.[5] tRNA는 아미노산 부착 부위와 안티코돈이라 불리는 부위를 갖는다. 안티코돈은 tRNA에 운반된 아미노산을 암호화하는 mRNA의 삼중항에 상보적인 RNA 삼중항이다.아미노아실 tRNA 합성효소는 특정 tRNA와 그 안티코돈 서열이 요구하는 아미노산 간의 결합을 촉매한다. 이 반응의 생성물은 아미노아실-tRNA이며, 아미노산은 카르복실기를 통해 tRNA의 3'-OH에 에스터 결합으로 결합한다. tRNA에 아미노산이 연결되면 tRNA는 "충전"된 것으로 간주된다. 박테리아에서 이 아미노아실-tRNA는 EF-Tu에 의해 리보솜으로 운반되며, 여기서 mRNA 코돈은 특정 tRNA 안티코돈에 상보적인 염기쌍을 통해 일치한다. tRNA를 잘못된 아미노산과 잘못 짝지은 아미노아실-tRNA 합성효소는 잘못 충전된 아미노아실-tRNA를 생성하여 단백질의 해당 위치에 부적절한 아미노산을 생성할 수 있다.[6]
2. 3. 번역 과정
단백질 생성의 기본 과정은 리보솜이 단백질의 끝에 한 번에 하나의 아미노산을 추가하는 것이다.[1] 추가할 아미노산의 유형은 전령 RNA(mRNA) 분자에 의해 결정된다. 추가되는 각 아미노산은 mRNA의 3개의 뉴클레오타이드 서브시퀀스(코돈)에 매칭된다. 각 코돈에 대해 해당 아미노산이 수용된다. 사슬에 추가된 연속적인 아미노산은 mRNA의 연속적인 뉴클레오타이드 삼중항에 매칭된다. 이러한 방식으로 템플릿 mRNA 사슬의 뉴클레오타이드 서열은 생성된 아미노산 사슬의 아미노산 서열을 결정한다.[3] 아미노산의 추가는 펩타이드의 C-말단에서 발생하며, 따라서 번역은 아민기에서 카복실기 방향으로 진행된다.[4]mRNA는 염색체에서 리보솜으로 리보뉴클레오타이드 서열로 인코딩된 유전 정보를 전달한다. 리보뉴클레오타이드는 코돈이라고 하는 일련의 뉴클레오타이드 삼중항에서 번역 기계에 의해 "읽혀진다". 각 삼중항은 특정 아미노산을 암호화한다.
리보솜은 리보솜 RNA(rRNA)와 단백질을 포함하는 다중 소단위체 구조이며, 아미노산이 단백질로 조립되는 "공장"이다. 전령 RNA(tRNA)는 아미노산을 리보솜으로 수송하는 작은 비코딩 RNA 사슬(74–93 뉴클레오타이드)이다. tRNA는 아미노산 부착 부위와 안티코돈 부위를 갖는다. 안티코돈은 그들의 화물인 아미노산을 암호화하는 mRNA 삼중항에 상보적인 RNA 삼중항이다.
아미노아실 tRNA 합성효소는 특정 tRNA와 그 안티코돈 서열이 요구하는 아미노산 간의 결합을 촉매한다. 이 반응의 생성물은 아미노아실-tRNA이다. tRNA에 아미노산이 연결되면 tRNA는 "충전"된 것으로 간주된다. 박테리아에서 이 아미노아실-tRNA는 EF-Tu에 의해 리보솜으로 운반되며, 여기서 mRNA 코돈은 특정 tRNA 안티코돈에 상보적인 염기쌍을 통해 일치한다. tRNA를 잘못된 아미노산과 잘못 짝지은 아미노아실-tRNA 합성효소는 잘못 충전된 아미노아실-tRNA를 생성하여 단백질의 해당 위치에 부적절한 아미노산을 생성할 수 있다. 이러한 유전 암호의 "잘못된 번역"[6]은 대부분의 유기체에서 자연적으로 낮은 수준으로 발생하지만, 특정 세포 환경은 허용적인 mRNA 해독을 증가시켜 때로는 세포에 이점을 준다.
리보솜에는 tRNA에 대한 두 개의 결합 부위가 있다. 아미노아실 부위(A)와 펩티딜 부위/출구 부위(P/E)이다. mRNA와 관련하여 세 부위는 5'에서 3' E-P-A로 정렬되는데, 이는 리보솜이 mRNA의 3' 말단을 향해 이동하기 때문이다. A 부위는 mRNA의 상보적인 코돈이 있는 들어오는 tRNA에 결합한다. P/E 부위는 성장하는 폴리펩타이드 사슬이 있는 tRNA를 유지한다. 아미노아실-tRNA가 처음에 mRNA의 해당 코돈에 결합하면 A 부위에 있다. 그런 다음 A 부위의 tRNA 아미노산과 P/E 부위에 충전된 tRNA의 아미노산 사이에 펩타이드 결합이 형성된다. 성장하는 폴리펩타이드 사슬은 A 부위의 tRNA로 전달된다. 전위가 발생하여 tRNA를 아미노산이 없는 P/E 부위로 이동시킨다. 폴리펩타이드 사슬로 이제 충전된 A 부위에 있던 tRNA는 P/E 부위로 이동하고 비충전 tRNA는 떠나고 다른 아미노아실-tRNA가 A 부위에 들어가서 과정을 반복한다.[7]
새로운 아미노산이 사슬에 추가된 후, tRNA가 리보솜 밖으로 방출되어 세포질로 들어간 후, 전위 효소 EF-G (세균) 및 a/eEF-2 (진핵생물 및 고세균)에 결합된 GTP의 가수분해에 의해 제공되는 에너지는 리보솜을 3' 말단을 향해 한 코돈씩 이동시킨다. 단백질의 번역에 필요한 에너지는 상당하다. ''n''개의 아미노산을 포함하는 단백질의 경우, 번역에 필요한 고에너지 인산 결합의 수는 4''n''-1이다.[8] 번역 속도는 다양하며, 진핵 세포(초당 최대 6~9개의 아미노산 잔기)보다 원핵 세포에서 훨씬 높다(초당 최대 17~21개의 아미노산 잔기).[9]
번역은 메신저 RNA(mRNA)가 핵 밖에 있는 리보솜에서 해독되어 특정 아미노산 사슬(폴리펩타이드)이 만들어지는 과정이다. 그 후, 폴리펩타이드는 활성단백질로 접히게 되어 세포 내에서 그 역할을 수행한다. 리보솜은 mRNA코돈에 상보적인 tRNA안티코돈 서열의 결합을 유도하여 해독을 진행한다.
번역은 다음 3단계로 진행된다.
1. '''개시''': 표적 mRNA 주위에 리보솜이 모인다. 첫 번째 tRNA가 mRNA의 개시 코돈에 결합한다.
2. '''신장''': 리보솜 소단위체에 의해 확인된(''수용'') tRNA는 운반한 아미노산을 리보솜 대단위체로 옮기고, 그 전에 수용된 아미노산에 결합한다(''펩티드 전이''). 그 후, 리보솜은 다음 mRNA 코돈으로 이동하여 프로세스를 계속하고(''전좌''), 아미노산 사슬을 형성한다.
3. '''종결''': 종결 코돈에 도달하면 리보솜은 폴리펩타이드를 방출한다. 리보솜 복합체는 그대로 남아, 다음에 번역될 mRNA로 이동한다.
원핵생물(세균 및 고세균)의 경우, 번역은 세포질 기질에서 일어나며, 리보솜의 대단위체와 소단위체가 mRNA에 결합한다. 진핵생물의 경우, 번역은 세포질 내 또는 소포체의 막을 넘어 공동 번역 전위라고 불리는 과정을 통해 일어난다. 공동 번역 전위에서는 리보솜과 mRNA의 복합체 전체가 조면 소포체 (ER)의 외막에 결합하고, 새로운 단백질이 합성되어 ER 내로 방출된다. 새로 만들어진 폴리펩타이드는 향후 소포 수송이나 세포 외부로의 분비를 위해 ER 내에 저장되거나, 즉시 분비된다.
3. 번역의 단계
번역은 개시, 신장, 종결의 세 단계로 진행된다.
# '''개시''': 표적 mRNA 주위에 리보솜이 모인다. 첫 번째 tRNA가 mRNA의 개시 코돈에 결합한다.
# '''신장''': 리보솜 소단위체에 의해 확인된 tRNA는 운반한 아미노산을 리보솜 대단위체로 옮기고, 그전에 수용된 아미노산에 결합한다(펩티드 전이). 그 후, 리보솜은 다음 mRNA 코돈으로 이동하여 과정을 계속하고(전좌), 아미노산 사슬을 형성한다.
# '''종결''': 종결 코돈에 도달하면 리보솜은 폴리펩타이드를 방출한다. 리보솜 복합체는 그대로 남아, 다음에 번역될 mRNA로 이동한다.[6]
원핵생물(세균 및 고세균)의 경우, 번역은 세포질 기질에서 일어나며, 리보솜의 대단위체와 소단위체가 mRNA에 결합한다. 진핵생물의 경우, 번역은 세포질 내 또는 소포체의 막을 넘어 공동 번역 전위라고 불리는 과정을 통해 일어난다. 공동 번역 전위에서는 리보솜과 mRNA의 복합체 전체가 조면 소포체 (ER)의 외막에 결합하고, 새로운 단백질이 합성되어 ER 내로 방출된다. 새로 만들어진 폴리펩타이드는 향후 소포 수송이나 세포 외부로의 분비를 위해 ER 내에 저장되거나, 즉시 분비된다.
폴리펩타이드의 종결은 리보솜의 A 부위가 mRNA 상의 종결 코돈(UAA, UAG, 또는 UGA)으로 채워질 때 발생하며, 단백질의 1차 구조를 생성한다. tRNA는 일반적으로 종결 코돈을 인식하거나 결합할 수 없다. 대신, 종결 코돈은 방출 인자 단백질[11]의 결합을 유도하여 리보솜의 펩티딜 전이 효소 중심[1]으로부터 폴리펩타이드 사슬의 가수 분해에 의해 전체 리보솜/mRNA 복합체의 해체를 촉진한다.[12] 약물 또는 mRNA 상의 특수 서열 모티프는 리보솜 구조를 변경하여 유사 인지 tRNA가 방출 인자 대신 종결 코돈에 결합하게 할 수 있다. 이러한 '번역 리드스루'의 경우, 리보솜이 다음 종결 코돈을 만날 때까지 번역이 계속된다.[13]
전이 RNA(tRNA), 리보솜 RNA(rRNA), 핵 내 저분자 RNA(snRNA) 등, 많은 종류의 전사 RNA는 단백질로 번역되지 않는다.
많은 항생제는 번역을 억제하여 작용한다. 예를 들어, 아니소마이신, 시클로헥시미드, 클로람페니콜, 테트라사이클린, 스트렙토마이신, 에리스로마이신, 퓨로마이신이 있다. 세균과 같은 원핵생물의 리보솜은 진핵생물의 리보솜과 구조가 다르기 때문에, 항생제는 진핵생물의 숙주 세포에 해를 끼치지 않으면서, 감염된 세균을 특이적으로 표적으로 할 수 있다.
3. 1. 개시 (Initiation)
리보솜의 작은 소단위체가 개시 인자의 도움을 받아 mRNA의 5' 말단에 결합한다. 세균과 소수의 고세균에서 단백질 합성 개시는 샤인-달가노 서열이라고 하는 mRNA 상의 퓨린이 풍부한 개시 서열을 인식하는 것을 포함한다. 샤인-달가노 서열은 30S 리보솜 소단위체의 16S rRNA 부분의 3' 말단에 있는 상보적인 피리미딘이 풍부한 서열에 결합한다. 이러한 상보적 서열의 결합은 30S 리보솜 소단위체가 mRNA에 결합되고, 개시 코돈이 P-부위의 30S 부분에 위치하도록 정렬되도록 한다. mRNA와 30S 소단위체가 적절하게 결합되면, 개시 인자가 개시 tRNA-아미노산 복합체, f-Met-tRNA를 30S P 부위로 가져온다. 개시 단계는 50S 소단위체가 30S 소단위체에 결합하여 활성 70S 리보솜을 형성하면 완료된다.[10]3. 2. 신장 (Elongation)
단백질 생성의 기본 과정은 단백질의 끝에 한 번에 하나의 아미노산을 추가하는 것이며, 이 작업은 리보솜에 의해 수행된다.[1] 추가할 아미노산 유형은 전령 RNA(mRNA) 분자에 의해 결정된다. 추가되는 각 아미노산은 mRNA의 3개의 뉴클레오티드 서브시퀀스에 매칭된다. 사슬에 추가된 연속적인 아미노산은 mRNA의 연속적인 뉴클레오티드 삼중항에 매칭된다. 이러한 방식으로 템플릿 mRNA 사슬의 뉴클레오티드 서열은 생성된 아미노산 사슬의 아미노산 서열을 결정한다.[3] 아미노산의 추가는 펩타이드의 C-말단에서 발생하며, 따라서 번역은 아민에서 카르복실 방향으로 진행된다.[4]리보솜에는 tRNA에 대한 두 개의 결합 부위가 있는데, 아미노아실 부위(약자 A)와 펩티딜 부위/출구 부위(약자 P/E)이다. mRNA와 관련하여 세 부위는 5'에서 3' E-P-A로 정렬되는데, 이는 리보솜이 mRNA의 3' 말단을 향해 이동하기 때문이다. A 부위는 mRNA의 상보적인 코돈이 있는 들어오는 tRNA에 결합한다. P/E 부위는 성장하는 폴리펩타이드 사슬이 있는 tRNA를 유지한다. 아미노아실-tRNA가 처음에 mRNA의 해당 코돈에 결합하면 A 부위에 있다. 그런 다음 A 부위의 tRNA 아미노산과 P/E 부위에 충전된 tRNA의 아미노산 사이에 펩타이드 결합이 형성된다. 성장하는 폴리펩타이드 사슬은 A 부위의 tRNA로 전달된다. 전위가 발생하여 tRNA를 아미노산이 없는 P/E 부위로 이동시킨다. 폴리펩타이드 사슬로 이제 충전된 A 부위에 있던 tRNA는 P/E 부위로 이동하고 비충전 tRNA는 떠나고 다른 아미노아실-tRNA가 A 부위에 들어가서 과정을 반복한다.[7]
새로운 아미노산이 사슬에 추가된 후, tRNA가 리보솜 밖으로 방출되어 세포질로 들어간 후, 전위 효소 EF-G (세균) 및 a/eEF-2 (진핵생물 및 고세균)에 결합된 GTP의 가수분해에 의해 제공되는 에너지는 리보솜을 3' 말단을 향해 한 코돈씩 이동시킨다. 단백질의 번역에 필요한 에너지는 상당하다. ''n''개의 아미노산을 포함하는 단백질의 경우, 번역에 필요한 고에너지 인산 결합의 수는 4''n''-1이다.[8] 번역 속도는 다양하며, 진핵 세포(초당 최대 6~9개의 아미노산 잔기)보다 원핵 세포에서 훨씬 높다(초당 최대 17~21개의 아미노산 잔기).[9]
3. 3. 종결 (Termination)
종결 코돈 (정지 코돈)에 도달하면 리보솜은 폴리펩타이드를 방출한다. 리보솜 복합체는 그대로 남아, 다음에 번역될 mRNA로 이동한다.[6]4. 번역의 오류와 조절
번역은 높은 정확도로 일어나지만, 오류가 발생할 수 있다. 번역 과정에서 오류가 발생하면 잘못된 단백질이 만들어지거나 번역이 일찍 중단될 수 있다. 이러한 오류는 tRNA가 잘못된 코돈에 결합하거나, tRNA에 잘못된 아미노산이 결합하면서 발생한다.
번역 과정은 진핵생물과 원핵생물 모두에서 고도로 조절된다. 번역 조절은 단백질 합성의 전체 속도에 영향을 미칠 수 있으며, 이는 세포의 대사 상태 및 증식 상태와 밀접하게 연관되어 있다.[18]
4. 1. 번역 오류
리보솜은 보통 정확하게 작동하지만, 번역 과정에서 오류가 발생하여 잘못된 단백질이 만들어지거나 번역이 일찍 중단될 수 있다. 이러한 오류는 tRNA가 잘못된 코돈에 결합하거나, tRNA에 잘못된 아미노산이 결합하면서 발생한다. 단백질 합성 오류율은 실험 조건에 따라 다르지만, 대략 105개에서 103개의 아미노산 중 1개꼴로 잘못 삽입되는 것으로 추정된다. 조기 번역 중단율은 번역된 코돈당 10−4 정도의 빈도로 발생하는 것으로 추정된다.4. 2. 번역 조절
번역 과정은 진핵생물과 원핵생물 모두에서 고도로 조절된다. 번역의 조절은 단백질 합성의 전체 속도에 영향을 미칠 수 있으며, 이는 세포의 대사 상태 및 증식 상태와 밀접하게 연관되어 있다.[18]이 복잡한 과정을 더 깊이 탐구하기 위해 과학자들은 일반적으로 리보솜 프로파일링이라는 기술을 사용한다.[18] 이 방법을 통해 연구자들은 번역체(번역이 진행 중인 mRNA와 리보솜의 복합체)를 스냅샷으로 찍어, 주어진 시간에 mRNA의 어떤 부분이 리보솜에 의해 단백질로 번역되는지 보여준다. 리보솜 프로파일링은 번역 역학에 대한 귀중한 통찰력을 제공하며, 유전자 서열, mRNA 구조 및 번역 조절 간의 복잡한 상호 작용을 밝혀낸다. 예를 들어, 이 방법을 활용한 연구에 따르면 유전적 차이와 그에 따른 mRNA로서의 발현 또한 RNA 특이적 방식으로 번역 속도에 영향을 미칠 수 있음이 밝혀졌다.[19]
최근 개발된 기술은 단일 세포 리보솜 프로파일링으로, 개별 세포 수준에서 번역 과정을 연구할 수 있게 해준다.[20] 이는 동일한 유형의 세포조차도 단백질 합성에 상당한 변동성을 보일 수 있다는 점에서 특히 중요하다. 단일 세포 리보솜 프로파일링은 세포의 이질적인 특성을 밝히는 데 기여하여, 번역 조절이 세포 행동, 대사 상태 및 다양한 자극 또는 조건에 대한 반응성에 어떻게 영향을 미칠 수 있는지에 대한 보다 미묘한 이해를 이끌어낼 수 있다.
5. 번역과 질병
번역 과정에 이상이 생기면 다양한 질병이 유발될 수 있다.
5. 1. 암
암세포는 번역 조절 기전을 이용하여 생존과 증식을 촉진한다. RAS-MAPK, PI3K/AKT/mTOR, MYC, WNT-β-카테닌 경로 등 주요 발암 경로는 번역을 통해 유전체를 재프로그래밍한다.[22] 암세포는 세포 스트레스에 적응하기 위해 번역을 조절하며, 스트레스 동안 세포는 스트레스를 완화하고 생존을 촉진할 수 있는 mRNA를 번역한다. 다양한 암에서 AMPK가 발현되는데, AMPK 활성화는 영양 결핍으로 유발된 세포 자멸사를 암세포가 회피하는 일련의 반응을 유발한다.[21]6. 번역의 수학적 모델링
번역 과정을 이해하기 위해 다양한 수학적 모델이 개발되었다. 번역은 다음과 같은 기본적인 과정을 거친다.[27]
# mRNA 분자의 생성(스플라이싱 포함)
# 개시 인자를 사용한 분자 개시 (원형화 단계를 포함할 수 있지만, 보편적으로 필요하지 않음)
# 번역 개시, 작은 리보솜 소단위체 모집
# 전체 리보솜 조립
# 신장 (mRNA를 따라 리보솜이 이동하며 단백질 생산)
# 번역 종결
# mRNA 분자 분해
# 단백질 분해
이러한 번역 과정은 오랫동안 다양한 물리적 모델의 대상이었다.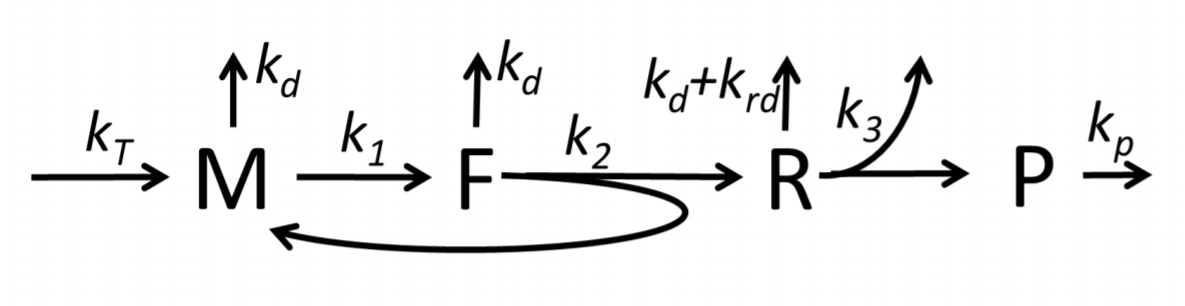
6. 1. 모델의 종류
번역 과정을 모델링하기 위해 다양한 기법들이 사용되어 왔다. 초기에는 아미노산 결합 과정에 대한 상세한 운동 모델과 컴퓨터 시뮬레이션이 주를 이루었다.[23] 지난 40년 동안, 화학 반응 속도론 기반의 단백질 합성 모델이 많이 개발되었다.[24][25]화학 반응 속도론 외에도, 전체 비대칭 단순 배제 과정(TASEP),[26] 확률적 부울 네트워크(PBN), 페트리넷, 최대 플러스 대수와 같은 모델링 기법이 단백질 합성의 동역학을 모델링하는 데 적용되었다.
단백질 합성의 기본 모델은 "유용한 모델은 단순하고 확장 가능하다"는 패러다임에 따라 개발되었다.[28] 가장 단순한 모델 ''M0''는 반응 운동 메커니즘으로 표현된다.
* M – 조립 중인 리보솜에 의해 점유되지 않은 번역 개시 부위가 있는 mRNA의 양,
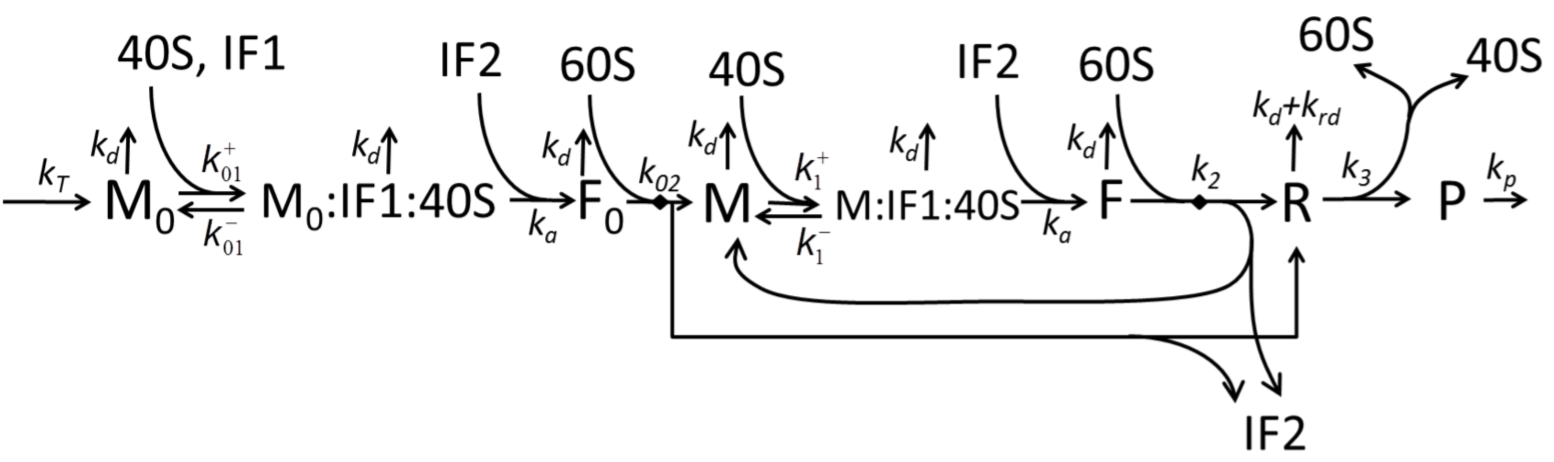
이 모델은 40S, 60S 및 개시 인자(IF) 결합을 포함하도록 확장되었다(그림 M1').[27] 또한, 마이크로RNA가 단백질 합성에 미치는 영향을 포함하도록 확장되었다.[29]
6. 2. 모델의 활용
번역에서 단백질을 만들기 위한 아미노산 구축 과정은 오랫동안 다양한 물리적 모델의 대상이었다.[23] 지난 40년 동안 단백질 합성에 대한 많은 화학적 운동 기반 모델이 개발 및 분석되었다.[24][25] 화학적 운동 외에도, 전체 비대칭 단순 배제 과정,[26] 확률적 부울 네트워크, 페트리 넷 및 최대 플러스 대수와 같은 다양한 모델링 형식이 단백질 합성 또는 일부 단계의 상세한 운동을 모델링하는 데 적용되었다."유용한 모델은 단순하고 확장 가능하다"는 패러다임을 따라,[28] 단백질 합성의 기본 모델이 개발되었다.[27] 가장 단순한 모델 ''M0''는 반응 운동 메커니즘으로 표현된다. 이 모델은 40S, 60S 및 개시 인자 (IF) 결합을 포함하도록 일반화되었다. 마이크로RNA가 단백질 합성에 미치는 영향을 포함하도록 더욱 확장되었다.[29] 이 계층 구조의 대부분의 모델은 분석적으로 해결할 수 있다. 이러한 솔루션은 다양한 특정 합성 조절 메커니즘의 '운동학적 특징'을 추출하는 데 사용되었다.
* M – 조립 중인 리보솜에 의해 점유되지 않은 번역 개시 부위가 있는 mRNA의 양,
7. 유전 암호
전령 RNA(mRNA)는 염색체에서 리보솜으로 리보뉴클레오티드 서열로 인코딩된 유전 정보를 전달한다. 리보뉴클레오티드는 코돈이라고 하는 일련의 뉴클레오티드 삼중항으로 번역된다. 각 코돈은 특정 아미노산을 암호화한다.
리보솜은 리보솜 RNA(rRNA)와 단백질을 포함하는 다중 소단위체 구조로, 아미노산을 단백질로 조립하는 "공장" 역할을 한다. 전이 RNA(tRNA)는 아미노산을 리보솜으로 수송하는 작은 비코딩 RNA 사슬(74–93 뉴클레오티드)이다. tRNA는 아미노산 부착 부위와 안티코돈 부위를 갖는다. 안티코돈은 tRNA에 결합된 아미노산을 암호화하는 mRNA 코돈에 상보적인 RNA 삼중항이다.
아미노아실 tRNA 합성효소는 특정 tRNA와 그 안티코돈 서열이 요구하는 아미노산 간의 결합을 촉매한다. 이 반응의 생성물은 아미노아실-tRNA이다. 아미노산은 카르복실기를 통해 tRNA의 3' OH기에 에스터 결합으로 결합한다. tRNA에 아미노산이 연결되면 tRNA는 "충전"된 것으로 간주된다.
새로운 아미노산이 사슬에 추가되면, tRNA는 리보솜 밖으로 방출되어 세포질로 들어간다. 이후 전위 효소 EF-G(세균) 및 a/eEF-2(진핵생물 및 고세균)에 결합된 GTP의 가수분해로 제공되는 에너지를 통해 리보솜은 3' 말단을 향해 한 코돈씩 이동한다.
짧은 염기서열의 경우 수동으로, 긴 염기서열의 경우 컴퓨터를 통해 번역하는 것도 가능하다. 이를 통해 생물학자와 화학자는 종이에 암호화된 단백질의 화학 구조를 그릴 수 있다.
DNA/RNA 서열을 단백질 서열로 번역할 수 있는 많은 컴퓨터 프로그램이 있다. 일반적으로 표준 유전 부호를 사용하여 수행되지만, 일부 프로그램은 대체 시작 코돈 사용과 같은 특수한 경우를 처리할 수 있다. 예를 들어, 드문 대체 시작 코돈 CTG는 시작 코돈으로 사용될 때 메티오닌을 암호화하고, 다른 모든 위치에서는 류신을 암호화한다.[30]
7. 1. 코돈
mRNA의 3개 염기 조합인 코돈은 특정 아미노산을 지정한다. 리보솜 분자는 이 코드를 특정 아미노산 서열로 번역한다. 리보솜은 리보솜 RNA(rRNA)와 단백질을 포함하는 다중 소단위체 구조이며, 아미노산이 단백질로 조립되는 "공장"과 같다.[1]전령 RNA(tRNA)는 아미노산을 리보솜으로 수송하는 작은 비코딩 RNA 사슬(74–93 뉴클레오티드)이다.[5] tRNA는 아미노산 부착 부위와 안티코돈이라고 하는 부위를 갖는다. 안티코돈은 그들의 화물인 아미노산을 암호화하는 mRNA 삼중항에 상보적인 RNA 삼중항이다.
아미노아실 tRNA 합성효소(효소)는 특정 tRNA와 그 안티코돈 서열이 요구하는 아미노산 간의 결합을 촉매한다. 이 반응의 생성물은 아미노아실-tRNA이다. tRNA에 아미노산이 연결되면 tRNA는 "충전"된 것으로 간주된다. 박테리아에서 이 아미노아실-tRNA는 EF-Tu에 의해 리보솜으로 운반되며, 여기서 mRNA 코돈은 특정 tRNA 안티코돈에 상보적인 염기쌍을 통해 일치한다. tRNA를 잘못된 아미노산과 잘못 짝지은 아미노아실-tRNA 합성효소는 잘못 충전된 아미노아실-tRNA를 생성하여 단백질의 해당 위치에 부적절한 아미노산을 생성할 수 있다.[6]
리보솜에는 tRNA에 대한 두 개의 결합 부위가 있다. 아미노아실 부위(약자 A)와 펩티딜 부위/출구 부위(약자 P/E)이다. mRNA와 관련하여 세 부위는 5'에서 3' E-P-A로 정렬되는데, 이는 리보솜이 mRNA의 3' 말단을 향해 이동하기 때문이다. A 부위는 mRNA의 상보적인 코돈이 있는 들어오는 tRNA에 결합한다. P/E 부위는 성장하는 폴리펩타이드 사슬이 있는 tRNA를 유지한다. 아미노아실-tRNA가 처음에 mRNA의 해당 코돈에 결합하면 A 부위에 있다. 그런 다음 A 부위의 tRNA 아미노산과 P/E 부위에 충전된 tRNA의 아미노산 사이에 펩티드 결합이 형성된다. 성장하는 폴리펩타이드 사슬은 A 부위의 tRNA로 전달된다. 전위가 발생하여 tRNA를 아미노산이 없는 P/E 부위로 이동시킨다. 폴리펩타이드 사슬로 이제 충전된 A 부위에 있던 tRNA는 P/E 부위로 이동하고 비충전 tRNA는 떠나고 다른 아미노아실-tRNA가 A 부위에 들어가서 과정을 반복한다.[7]
7. 2. 개시 코돈과 종결 코돈
AUG는 메티오닌을 암호화하는 개시 코돈이며, 번역 시작을 알린다.[42] UAA, UAG, UGA는 종결 코돈이며, 번역 종결을 유도한다.[12]단백질 합성의 개시 과정은 리보솜의 작은 소단위체가 개시 인자(IF)의 도움을 받아 mRNA의 5' 말단에 결합하는 것을 포함한다. 세균과 소수의 고세균에서 단백질 합성 개시는 샤인-달가노 서열이라고 하는 mRNA 상의 퓨린이 풍부한 개시 서열을 인식하는 것을 포함한다. 샤인-달가노 서열은 30S 리보솜 소단위체의 16S rRNA 부분의 3' 말단에 있는 상보적인 피리미딘이 풍부한 서열에 결합한다. 이러한 상보적 서열의 결합은 30S 리보솜 소단위체가 mRNA에 결합되고, 개시 코돈이 P-부위의 30S 부분에 위치하도록 정렬되도록 한다. mRNA와 30S 소단위체가 적절하게 결합되면, 개시 인자가 개시 tRNA-아미노산 복합체, f-Met-tRNA를 30S P 부위로 가져온다. 개시 단계는 50S 소단위체가 30S 소단위체에 결합하여 활성 70S 리보솜을 형성하면 완료된다.[10]
폴리펩타이드의 종결은 리보솜의 A 부위가 mRNA 상의 종결 코돈(UAA, UAG, 또는 UGA)으로 채워질 때 발생하며, 단백질의 1차 구조를 생성한다. tRNA는 일반적으로 종결 코돈을 인식하거나 결합할 수 없다. 대신, 종결 코돈은 방출 인자 단백질[11](RF1 & RF2)의 결합을 유도하여 리보솜의 펩티딜 전이 효소 중심[1]으로부터 폴리펩타이드 사슬의 가수 분해에 의해 전체 리보솜/mRNA 복합체의 해체를 촉진한다.[12]
7. 3. 유전 암호의 변형
표준 유전 암호 외에 변형된 유전 암호가 발견되기도 한다. 미토콘드리아나 일부 생물 종에서는 표준 유전 암호와 다른 암호를 사용한다.[30] 예를 들어, 드문 대체 시작 코돈 CTG는 시작 코돈으로 사용될 때 메티오닌을 암호화하고, 다른 모든 위치에서는 류신을 암호화한다.[30]8. 번역표
효모 게놈과 같은 일반적인 진핵생물 서열을 다룰 때조차도, 특히 미토콘드리아 유전자의 번역을 위해 대체 번역표를 사용하는 것이 종종 필요하다. 현재 NCBI 분류군에 의해 GenBank의 서열 번역을 위해 다음과 같은 번역표가 정의되어 있다.[30]
참조
[1]
논문
The Peptidyl Transferase Center: a Window to the Past
2021-11
[2]
서적
Biology
McGraw Hill Education
[3]
서적
Biology
The Benjamin/Cummings Publishing Company
[4]
서적
Biochemistry
W. H. Freeman and Company
[5]
논문
The Evolution of tRNA Copy Number and Repertoire in Cellular Life
2023
[6]
논문
Mistranslation of the genetic code
2014-11
[7]
서적
Introduction to Genetic Analysis
W.H. Freeman and Company
[8]
웹사이트
Computational Analysis of Genomic Sequences utilizing Machine Learning
https://scholar.goog[...]
2022-01-12
[9]
논문
Growth-rate-dependent adjustment of ribosome function in chemostat-grown cells of the fungus Mucor racemosus
1982-02
[10]
논문
Mechanisms of the initiation of protein synthesis: in reading frame binding of ribosomes to mRNA
2011-02
[11]
논문
Global analysis of translation termination in E. coli
2017-03
[12]
논문
Stop codon recognition and interactions with peptide release factor RF3 of truncated and chimeric RF1 and RF2 from Escherichia coli
2003-12
[13]
논문
Functional Translational Readthrough: A Systems Biology Perspective
2016-08
[14]
논문
Errors in translational decoding: tRNA wobbling or misincorporation?
2019-03
[15]
논문
Evolutionary optimization of speed and accuracy of decoding on the ribosome
2011-10
[16]
논문
Quantitative assessment of ribosome drop-off in E. coli
2016-04
[17]
논문
A data-driven estimation of the ribosome drop-off rate in S. cerevisiae reveals a correlation with the genes length
2024-04
[18]
논문
Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling
2009-04
[19]
논문
Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans
2015-11
[20]
논문
Single-cell quantification of ribosome occupancy in early mouse development.
[21]
논문
The Role of Translation Control in Tumorigenesis and Its Therapeutic Implications
2020-03
[22]
논문
New frontiers in translational control of the cancer genome
2016-04
[23]
논문
Kinetics of biopolymerization on nucleic acid templates
1968
[24]
논문
Mathematical modelling of translation of mRNA in eucaryotes; steady state, time-dependent processes and application to reticulocytes
1980-09
[25]
논문
Dynamic model of the process of protein synthesis in eukaryotic cells
2007-01
[26]
논문
Dynamic model of the process of protein synthesis in eukaryotic cells
2007-01
[27]
논문
Basic, simple and extendable kinetic model of protein synthesis
2019-07
[28]
논문
Reply to Baveye and Darnault: Useful models are simple and extendable
2017-04
[29]
논문
Kinetic signatures of microRNA modes of action
2012-09
[30]
웹사이트
The Genetic Codes
https://www.ncbi.nlm[...]
National Center for Biotechnology Information (NCBI)
2022-05-31
[31]
논문
The Peptidyl Transferase Center: a Window to the Past
2021-11
[32]
서적
Biology
McGraw Hill Education
[33]
서적
Biology
The Benjamin/Cummings Publishing Company
[34]
서적
Biochemistry
W. H. Freeman and Company
[35]
논문
Mistranslation of the genetic code
2014-11
[36]
서적
Introduction to Genetic Analysis
W.H. Freeman and Company
[37]
웹사이트
Computational Analysis of Genomic Sequences utilizing Machine Learning
https://scholar.goog[...]
2022-01-12
[38]
논문
Growth-rate-dependent adjustment of ribosome function in chemostat-grown cells of the fungus Mucor racemosus
1982-02
[39]
논문
Errors in translational decoding: tRNA wobbling or misincorporation?
2019-03
[40]
논문
Evolutionary optimization of speed and accuracy of decoding on the ribosome
2011-10
[41]
논문
Quantitative assessment of ribosome drop-off in E. coli
2016-04
[42]
논문
Mechanisms of the initiation of protein synthesis: in reading frame binding of ribosomes to mRNA
2011-02
[43]
논문
Global analysis of translation termination in E. coli
2017-03
[44]
논문
Stop codon recognition and interactions with peptide release factor RF3 of truncated and chimeric RF1 and RF2 from Escherichia coli
2003-12
[45]
논문
Functional Translational Readthrough: A Systems Biology Perspective
2016-08
[46]
논문
Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans
2015-11
[47]
논문
The Role of Translation Control in Tumorigenesis and Its Therapeutic Implications
2020-03
[48]
논문
New frontiers in translational control of the cancer genome
2016-04
[49]
논문
Kinetics of biopolymerization on nucleic acid templates
1968
[50]
논문
Mathematical modelling of translation of mRNA in eucaryotes; steady state, time-dependent processes and application to reticulocytes
1980-09
[51]
논문
Dynamic model of the process of protein synthesis in eukaryotic cells
2007-01
[52]
논문
Dynamic model of the process of protein synthesis in eukaryotic cells
2007-01
[53]
논문
Reply to Baveye and Darnault: Useful models are simple and extendable
2017-04
[54]
논문
Basic, simple and extendable kinetic model of protein synthesis
2019-07
[55]
논문
Kinetic signatures of microRNA modes of action
2012-09
[56]
웹사이트
The Genetic Codes
https://www.ncbi.nlm[...]
アメリカ国立生物工学情報センター
2022-05-31
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com